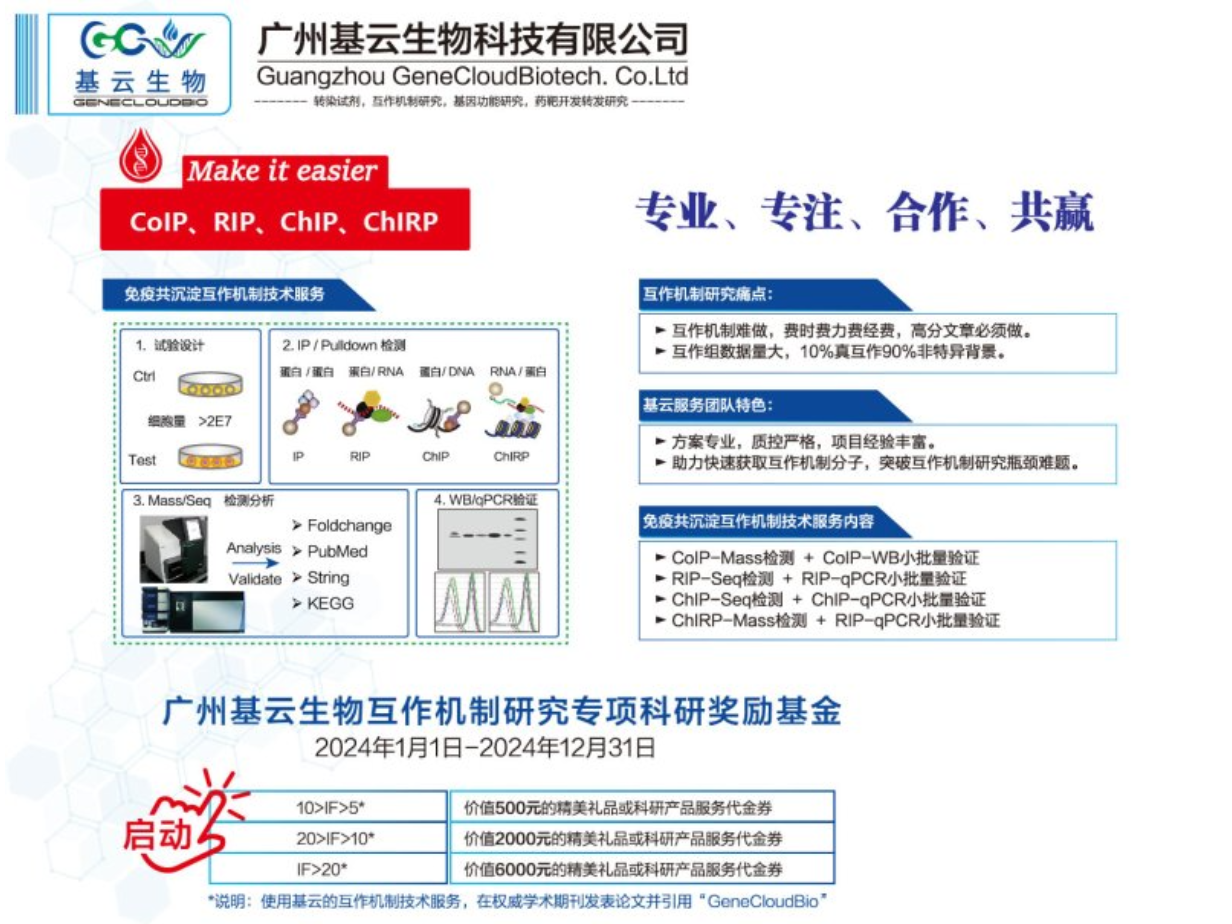
ChIP实验主要分为ChIP-qPCR和ChIP-seq两大类。ChIP-qPCR是一种结合了染色质免疫沉淀(ChIP)与实时荧光定量PCR(qPCR)的技术。它用于检测特定蛋白质(如转录因子)与特定DNA序列的结合情况,通过ChIP富集与目的蛋白结合的DNA片段,随后用qPCR技术对这些片段进行定量检测,以验证蛋白质与特定基因区域的结合关系。ChIP-qPCR适用于已知蛋白质与靶序列相互作用的研究,具有较高的灵敏度和特异性。而ChIP-seq则结合了ChIP与高通量测序技术,在全基因组范围内检测与特定蛋白质结合的DNA区域。该技术可以绘制出转录因子等蛋白质在全基因组范围内的结合位点图谱,对于未知靶序列的研究尤为重要。ChIP-seq能够提供更完整、高分辨率的结合信息,是探索转录调控网络、表观遗传机制等领域的有力工具。总的来说,ChIP-qPCR和ChIP-seq都是研究蛋白质与DNA相互作用的重要技术,选择使用哪种技术取决于研究的具体目标和需求。ChIP实验优点和缺点是什么。染色质免疫共沉淀ChIP-PCR

使用ChIP-seq快速确定下游靶标涉及多个关键步骤:首先,进行ChIP实验以富集与目标蛋白(如转录因子)结合的DNA片段。在这一步中,确保使用高质量的抗体以特异性地捕获目标蛋白与DNA的复合物。接着,将富集的DNA片段进行高通量测序。测序产生的数据将提供全基因组范围内目标蛋白的结合位点信息。然后,对测序数据进行生物信息学分析。这包括将测序读段比对到参考基因组上,识别并注释峰值区域,这些峰值区域表示目标蛋白与DNA的潜在结合位点。接下来,分析峰值区域在基因组中的分布,以确定下游靶标。特别关注那些位于基因启动子、增强子等调控区域的峰值,因为这些区域通常与基因表达调控密切相关。此外,还可以整合其他组学数据(如转录组学、表观遗传学数据等),以进一步验证和解释目标蛋白与下游靶标之间的调控关系。另外,通过实验验证(如qPCR、基因敲除或过表达等)来确认下游靶标的功能和调控作用。综上所述,通过ChIP-seq实验结合生物信息学分析和实验验证,可以快速而准确地确定下游靶标,并揭示目标蛋白在基因表达调控网络中的作用机制。内蒙古ChIP-SeqChIP-seq与ChIP-qPCR都应用于研究蛋白质在基因组上的结合情况。
随着技术的不断发展和完善,ChIP技术在未来研究中的应用前景将更加广阔。一方面,随着高通量测序技术的不断进步,我们可以获得更加系统、深入的蛋白质与DNA相互作用信息。另一方面,随着生物信息学方法的不断发展,我们可以对ChIP数据进行更加深入的分析和挖掘,从而揭示更多关于转录调控机制的信息。此外,随着单细胞测序技术的发展,我们可以进一步探索单细胞内蛋白质与DNA的相互作用模式,为揭示生命活动的奥秘提供更加深入的理解。
真核生物的基因组DNA以染色质的形式存在。因此,研究蛋白质与DNA在染色质环境下的相互作用是阐明真核生物基因表达机制的基本途径。染色质免疫沉淀技术(chromatin immunoprecipitation assay, CHIP)是目研究体内DNA与蛋白质相互作用的方法。它的基本原理是在活细胞状态下固定蛋白质-DNA复合物,并将其随机切断为一定长度范围内的染色质小片段,然后通过免疫学方法沉淀此复合体,特异性地富集目的蛋白结合的DNA片段,通过对目的片断的纯化与检测,从而获得蛋白质与DNA相互作用的信息。ChIP实验技术原理是什么。
ChIP-seq与ChIP-qPCR在实验技术、分辨率和数据分析方面存在明显的不同之处。首先,ChIP-seq结合了高通量测序技术,能够在全基因组范围内检测蛋白质与DNA的结合位点。它通过测序仪对富集的DNA片段进行大规模并行测序,生成海量的数据,从而提供高分辨率的结合位点信息。相比之下,ChIP-qPCR则侧重于对特定基因或基因区域进行定量分析,它通过荧光定量PCR技术检测富集的DNA片段的数量,具有更高的灵敏度和特异性,但只能针对已知序列进行分析。其次,ChIP-seq在分辨率上优于ChIP-qPCR。由于ChIP-seq可以对全基因组进行测序,它能够检测到更多的结合位点,包括那些低丰度或远离转录起始位点的结合事件。而ChIP-qPCR则受限于所选择的基因或基因区域,可能无法全局反映蛋白质在基因组上的结合情况。在数据分析方面,ChIP-seq生成的数据需要进行复杂的生物信息学分析,包括序列比对、峰值调用、注释和富集分析等步骤。而ChIP-qPCR的数据分析相对简单,主要通过比较不同样品间的荧光信号强度来判断蛋白质的结合情况。使用ChIP-seq快速确定下游靶标涉及多个关键步骤。DNA蛋白互作检测ChIP-Sequence
ChIP-seq实验有哪些有点。染色质免疫共沉淀ChIP-PCR
ChIP-Seq检测原理:ChIP-Seq检测原理和RIP-Seq类似,不同的是前者利用目的蛋白抗体将相应的DNA-蛋白复合物沉淀下来,然后分离纯化捕获DNA,结合高通量测序技术对目标DNA进行测序分析。ChIP-Seq服务要点和RIP-Seq类似,精简如下:(1)试验设计:同RIP-Seq。(2)蛋白表达和细胞量:比RIP-Seq细胞用量要求大,建议不少于10e7(金标准:320g离心沉淀100ul)。(3)抗体关键质控:同IP-Mass和RIP-Seq。(4)IP送样建议:细胞培养好后,收集前,先进行交联,再收样冻存。(5)互作DNA筛选和验证:同RIP-Seq。ChIP-Seq优劣势:优势:高通量获得目的蛋白的专属DNA互作库。劣势:技术门槛高,一般需要整包交给专业的服务商开展检测。ChIP-Seq应用扩展:(1)蛋白DNA相互作用数据,是探究转录调控机制研究的重要内容,体现机制研究的深度,能显著提高临床基础类研究文章的档次。(2)蛋白DNA互作组检测,常用于蛋白的转录调控研究,如转录因子,转录调控蛋白等。(3)蛋白DNA互作,其结合DNA的区域,是进一步研究互作机制和功能的关键内容,能够显著提高机制研究的高度。染色质免疫共沉淀ChIP-PCR