- 品牌
- 南京庚乾信息科技有限公司
- 型号
- 生命科学软件
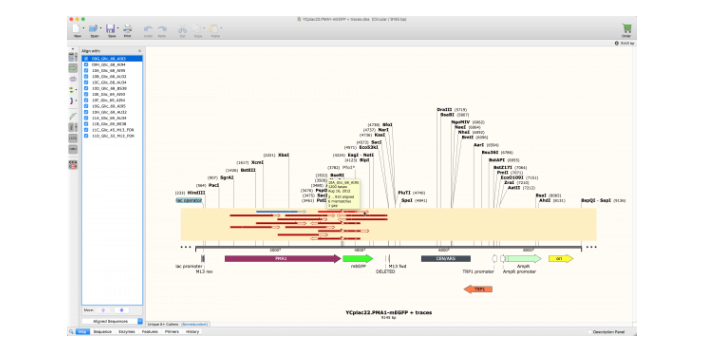
自动查看质粒特征-生命科学软件•使用SnapGene的精选特征数据库或您自己的自定义特征注释您的质粒特征•在质粒图谱上或在序列视图上详细显示酶位点、特征、引物、ORF、翻译等•使用灵活的注释和可视化控件自定义您的地图使用比对工具检查您的序列-生命科学软件•使用强大的对齐参考工具验证您的测序结构是否与您的模拟结构匹配•使用可靠的算法对齐序列以进行成对和多重对齐,包括ClustalOmega、MAFFT、MUSCLE和T-Coffee•使用CAP3将Sanger测序读入完整的重叠群适用于生命科学行业的控制系统和软件。山东数据分析生命科学软件下载
从Star开始CSD序列前25个左右碱基,此时注意温度比较好在60度左右(22-40碱基数目之间都可以),点击Primers>addprimer>topstrand,点OK。-生命科学软件从END开始CSD序列后25个左右碱基,此时注意温度比较好在60度左右(22-40碱基数目之间都可以),但也要兼顾碱基的长度,如果太长则不好扩增PCR。点击Primers>addprimer>bottomstrand,点OK.这一步先做到这,后续再添加酶和碱基。-生命科学软件点击Emazy>chooseemazy>先移除右边框中的酶,然后再添加你所在实验室中已有的酶,我在这里随便选了6种。如下图所示,然后点确定。北京数据分析生命科学软件安装生物医学科研必备神器一览。
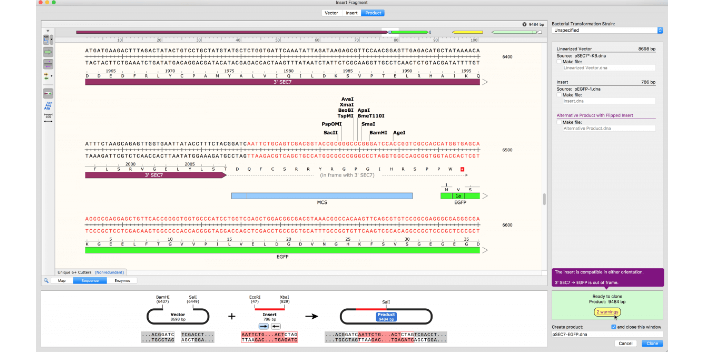
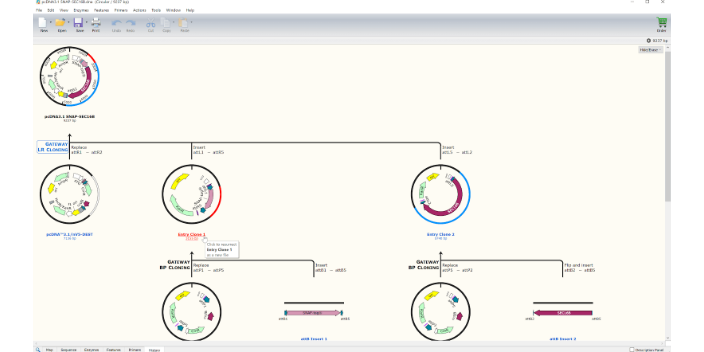
载体构建——同源重组法-生命科学软件。还是以基因ATG7为例,首先在NCBI,TAIR等基因网站找到这个基因,复制CDS序列。将序列插入newdnafile,然后重命名文件。选中全长后,点击Features>addfeature>labelname改为CDS>type改为CDS。这样的目的是为了方便后续检测基因是否插入成功。查看实验室已有的酶,点击Emazy>chooseemazy>先移除右边框中的酶,然后再添加你所在实验室中已购买的酶。保存文件为ATG文件。后续要在载体中插入到这个文件所以要先保存。-生命科学软件。打开载体文件,找到到多酶切位点序列。查看哪些酶切位点可用且不会切到基因片段,然后确定酶切位点,可用一个酶,也可以用两个酶,不过比较好选两个酶。在载体文件中选择要两个酶切位点,点击Action>In-fusioncloning>Insertfragment。
SnapGene和SnapGeneViewer进入的主界面上是一样的,区别在于SnapGene要比Viewer多不少可用功能,也就是Viewer上有SnapGene图标标示的都是需要付费版才能用的,此外SnapGene现在也有中文版了。-生命科学软件两个版本比较大的区别就在于行动和工具两个工具栏中,SnapGene多了很多PCR模拟,BLAST还有琼脂糖凝胶模拟等功能,但是基础的质粒图谱查看SnapGeneViewer还是能够满足的。水平图谱查看界面-生命科学软件序列界面与图谱界面类似,不同的是左侧的功能栏。多出了显示小图谱,氨基酸设置和压缩格式等。只是在显示中与区别,可以自行去点一下,看看每一项有什么区别。生命科学软件行业研究分析报告。
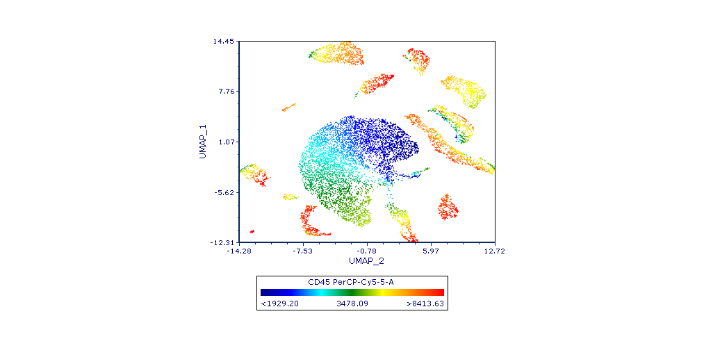
在生命科学领域,软件扮演着至关重要的角色,从数据收集、分析到管理、可视化,再到模拟和解释,每个阶段都有其独特工具。下面是一些生命科学软件的测评概览,涵盖不同方面,旨在帮助你了解其优势、局限和适用场景:1. 序列分析软件 - BLASTIGN (Basic Local Alignment Search Tool)•优势:**经典且强大的序列比对算法,广泛应用于基因序列搜索和比对。•局限:处理大数据集时速度可能较慢,图形界面不够现代化。•适用场景:适合于基础的序列比对和研究,尤其在教育和小规模数据集上。2. 基因组学工具 - GATKrona•优势:高效、高通量数据处理,支持复杂的基因组数据分析管道。•局限:学习曲线陡峭,需要一定的生物信息学背景。•适用场景:大规模基因组测序数据分析,如**研究、遗传病研究。3. 蛋白质结构与模拟 - PyMol•优势:强大的建模招建模与预测能力,支持多种分析。•局限:对计算资源要求高,复杂的模拟可能需高性能计算机。•适用场景:蛋白质结构研究、药物设计、分子动力学模拟。Mesquite下载-Mesquite官方版下载[生物学软件]。上海正版生命科学软件哪里有
生命科学软件有什么特点。山东数据分析生命科学软件下载
得到的结果如下图所示。下图中显示的酶是能够切断目的基因的酶,因此要排除。所以我们剩下的可以选择的酶包括Nde1,EcoR1,Pst1共3种酶。下一步打开载体DNA文件。-生命科学软件打开载体以pGADT7AD为例,查看切入位点的酶有哪些。在筛选的过程中还要注意酶切位点不能把基因切断。因此在选择酶切位点时候要保证两点。1.载体中多克隆位点中包含该限制性内切酶。2.目标基因可酶切的位置不包含该限制性内切酶。点击图中标记的地方MCS(多克隆位点)插入基因的位置。-生命科学软件。山东数据分析生命科学软件下载
Snapgene构建载体—酶切位点法-生命科学软件本文以拟南芥相关基因ATG7为例,打开Tair网页(专门的拟南芥基因库网站),找到基因那一栏,输入目标基因后得到结果。从图中可以看到描述这一栏写的这个基因的名称,其中就包括了ATG7。确认基因后点击目标基因。-生命科学软件找到SequenceRNAData:点击fulllengthCDS.打开页面后,复制全部序列。打开snapgene,点击newdnafile。黏贴复制文件,并重命名DNA文件,点击OK。选择全部序列后,点击Features>addfeature>labelname(CDS),type(CDS),点击OK。生命科学!SnapGe...
- 广东图像处理生命科学软件是什么 2024-05-20
- 湖南生命科学软件价格 2024-05-20
- 四川图表制作生命科学软件多少钱 2024-05-19
- 湖南文档处理生命科学软件哪里有 2024-05-19
- 广东自动化生命科学软件哪家好 2024-05-19
- 江苏GraphPad生命科学软件推荐 2024-05-19
- 苏州GraphPad Prism生命科学软件多少钱 2024-05-19
- 成都FCS Express生命科学软件哪个好 2024-05-19
- 深圳数据分析生命科学软件 2024-05-19
- 山东文档处理生命科学软件 2024-05-19
- 杭州数据分析生命科学软件多少钱 2024-05-19
- 福建FCS Express生命科学软件试用 2024-05-19
- 苏州Geneious Prime生命科学软件下载 2024-05-19
- 北京生命科学软件多少钱 2024-05-19
- 湖南Geneious Prime生命科学软件价格 2024-05-19
- 广东数据可视化处理生命科学软件 2024-05-18


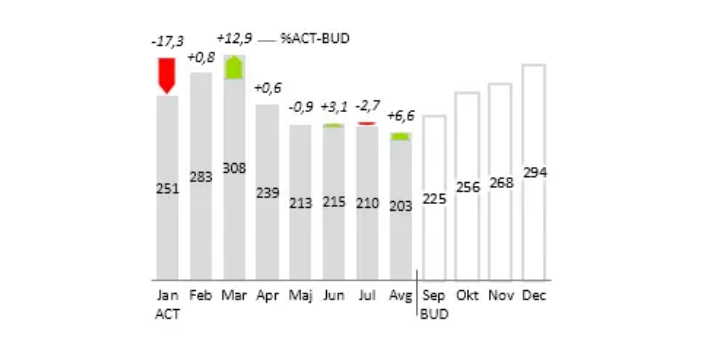
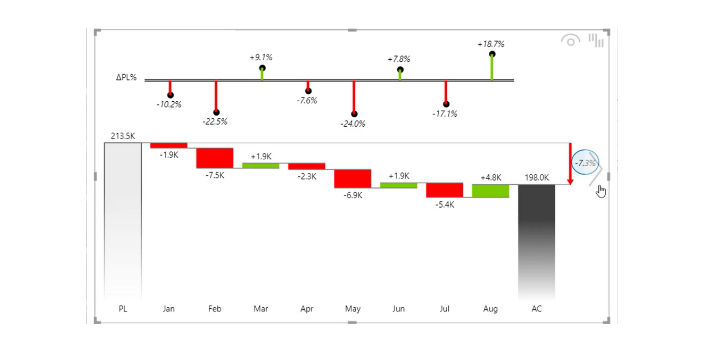
- 浙江数据库ZebraBI业务图表 06-08
- 江西了解ZebraBI帮助测试 06-08
- 上海汉化版DevExpress怎么样 06-08
- 湖南供应ZebraBI帮助测试 06-08
- 标准版DevExpress版本区别 06-08
- 浙江使用简单ZebraBI优化 Power BI 报告 06-08
- 湖南可视化数据ZebraBI官网 06-08
- 苏州Excel版ZebraBI插件 06-08
- 浙江专业级RAWZebraBI官网 06-08
- 浙江完整版DevExpress是什么软件 06-08